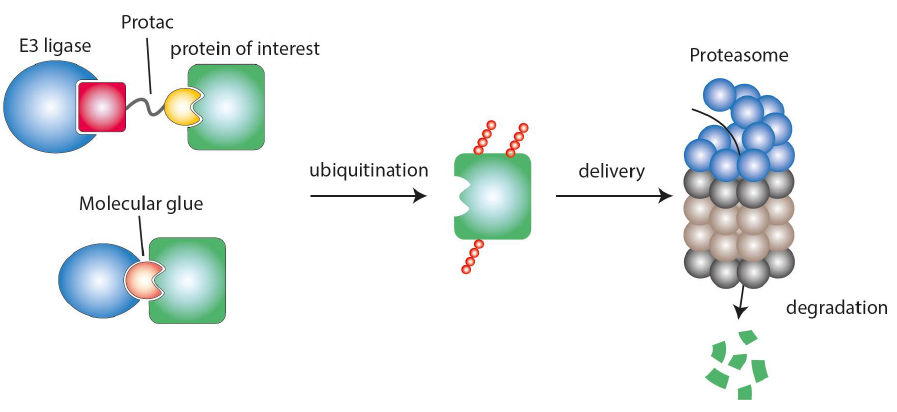
Ziel der Plattform ist es, die Wirkstoffklasse der proximity inducing drugs (PiDs) für die Behandlung immunvermittelter Erkrankungen nutzbar zu machen. PiDs sind bifunktionale Moleküle, die durch transiente Bindung an zwei Zielstrukturen diese in räumliche Nähe zueinander bringen und dadurch einen biologischen Effekt auslösen. Bei PiDs im engeren Sinne ist eine der beiden Strukturen eine Ubiquitin-E3-Ligase, die andere das Zielprotein, das durch diese E3-Ligase für den Abbau im Proteasom markiert wird. Diese PiDs führen damit zum vollständigen Verlust des Zielproteins mit all seinen, z.B. katalytischen, strukturgebenden oder regulatorischen Funktionen, was PiDs von klassischen Inhibitoren unterscheidet.
80% des humanen Proteoms gilt als »undruggable«
Trotz eines großen Arsenals an verfügbaren Wirkstoffklassen gelten ca. 80% des humanen Proteoms als therapeutisch nicht oder nur schwer zugänglich. Ein aktives Zentrum ist für die Wirkung von PiDs nicht erforderlich – damit wird es möglich, bisher als undruggable geltende, krankheitsrelevante Zielproteine zu adressieren.
Bei den PiDs, speziell den PROTACs (proteolysis-targeting chimeras) handelt es sich um eine neue, aber sehr intensiv beforschte Klasse von Molekülen. Die am weitesten fortgeschrittenen Wirkstoffe dieser Klasse befinden sich aktuell in frühen Phasen der klinischen Entwicklung, vor allem für onkologische Erkrankungen.
Charakterisierung und Optimierung von PiDs
Die Arbeiten innerhalb der Plattform sollen dazu dienen, die Infrastruktur, Prozesse und Arbeitsabläufe für das Design, die Synthese, die Charakterisierung und Optimierung von PiDs in in einer institutsübergreifenden Zusammenarbeit zu etablieren. Dabei sollen zwei Klassen von PiDs bearbeitet werden: PROTACS und LYTACs (lysosomal-targeting chimeras). Am Beispiel von einem oder mehreren ausgewählten Zielproteinen sollen PiDs identifiziert, ihre Wirksamkeit in Modellen immuno-inflammatorischer Erkrankungen geprüft und ihre Wirkungsweise mit der klassischer Inhibitoren verglichen werden.
 Fraunhofer Cluster of Excellence Immune-Mediated Diseases
Fraunhofer Cluster of Excellence Immune-Mediated Diseases